- Bestandteile des Reagenzienkits
| Spezifikationen | 50T | 100T |
| Katze. NEIN. | SN0201 | SN0202 |
| DNA-Extraktionssäulen (Satz) | 50 (Satz) | 100 (Satz) |
| Reagenzpufferlösung A | 30 ml | 2 × 30 ml |
| Reagenzpufferlösung C | 30 ml | 2 × 30 ml |
| Waschpuffer 1 | 15 ml | 2 × 15 ml |
| RNase A | 1ml | 1ml |
| Elutionspuffer | 20 ml | 20 ml |
| Bedienungsanleitung | 1 | 1 |
- Lagerung
Dieses Kit sollte bei Raumtemperatur gelagert werden (15-25℃) trocken und lagerfähig 12 Monate. DNA-Extraktions-Reinigungssäulen können in einer kühlen und trockenen Umgebung bis zu gelagert werden 1 Jahr. RNase A enthält ein Konservierungsmittel und kann bei Raumtemperatur transportiert werden, aber zur Langzeitlagerung, es sollte bei -20℃ gehalten werden.
- Anweisungen zur Verwendung des Reagenzienkits
3.1 Dieses Kit ist für molekularbiologische Forschungszwecke bestimmt und sollte nicht zur Diagnose oder Behandlung von Krankheiten verwendet werden.
3.2 Einige Bestandteile des Kits enthalten Reizstoffe; Es ist ratsam, die erforderlichen Vorsichtsmaßnahmen zu treffen (wie das Tragen von Schutzkleidung und Schutzbrillen).
3.3 Die Verwendung dieses Kits erfordert zusätzliche Ausrüstung wie eine Hochgeschwindigkeitszentrifuge, Wasserbad (Metallbad), Vortexmixer, Wasserfreies Ethanol, flüssiger Stickstoff, Chloroform, steriles entionisiertes Wasser, und EP-Röhren.
- Einführung in das Reagenzienkit
Der CTAB-basierte Anlage DNA-Reinigung Das Kit bietet eine verbesserte CTAB-Methode zur Reinigung von DNA, Verwendung eines spezifischen Bindungspuffers, der DNA effizient ausfällt, und sammelt anschließend hochreine DNA über eine Adsorptionssäule.
Dieses Kit wird häufig für Pflanzengewebe und Pilze verwendet, ist in der Lage, die gesamte DNA aus darin enthaltenen Proben zu extrahieren 2 Std. (einschließlich mitochondrialer DNA und Chloroplasten-DNA). Die extrahierte DNA kann direkt für nachgelagerte Experimente verwendet werden, z PCR, Southern Blot, und andere.
- Experimentelle Prinzipien und Verfahren
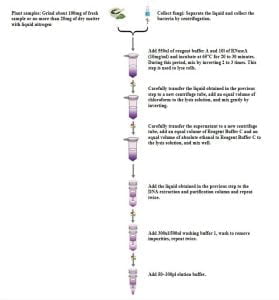
- Extraktionsprozess
Vorsichtsmaßnahmen vor Beginn des Experiments:
- Reagenzpuffer A und C kann bei niedrigen Temperaturen ausfallen. Es wird empfohlen, es auf 65°C zu erhitzen 5 Minuten einweichen und verwenden, nachdem sich die Niederschläge aufgelöst haben.
- WaschenPuffer 1 Es sollte die angegebene Menge an wasserfreiem Ethanol hinzugefügt werden, wie auf dem Flaschenetikett angegeben. Markieren Sie das Etikett, sobald Ethanol hinzugefügt wurde.
- Elutionspuffer ist ein 0.1x TE-Lösungmit minimalem EDTA-Gehalt. Wenn EDTA nachfolgende Experimente beeinflusst, Als Ersatz für den Elutionspuffer wird steriles entionisiertes Wasser empfohlen.
- Probenhandhabung:
- Materialsammlung und -lagerung:
Frisch gesammeltes Material, wenn nicht sofort verwendet, sollten in flüssigen Stickstoff gegeben und schließlich bei -80 °C gelagert werden. Getrocknete Materialien können bei Raumtemperatur gelagert werden.
- Wenn möglich, Sammeln Sie frisches Material, da es weniger Polysaccharide und Polyphenole enthält.
- Beim Sammeln von Pilzen aus Flüssigkultur, Trennen Sie die Flüssigkeit durch Zentrifugieren und sammeln Sie die Pilzkörper.
- Schleifen Sie herum 100 mg frischer Proben oder nicht mehr als 20 mg Trockenmaterial mit flüssigem Stickstoff.
(Notiz: Unterschiedliche Probenmengen erfordern möglicherweise eine Optimierung durch Vorversuche vor der Verwendung.)
- Hinzufügen 550 μl Reagenzpuffer A und 10 μl RNase A (10 mg/ml) um sicherzustellen, dass sich in der gemahlenen Probe keine Gewebeklumpen befinden. Gewebeklumpen sind schwer zu lysieren und können die DNA-Ausbeute verringern. Nicht mischen Reagenzpuffer A und RNase Avor dem Gebrauch.
- Bei 65 °C inkubieren 20-30 Protokoll, vorsichtig umdrehen 2-3 mal. Dieser Schritt dient der Zelllyse.
- Zentrifugieren Sie das Lysat 5 Minuten um 14,000 U/min (20,000×g).
(Notiz: Einige Pflanzenmaterialien können in diesem Schritt viele klebrige Substanzen enthalten, die DNA in nachfolgenden Schritten scheren kann. Ideal, Entfernen Sie diese Substanzen, indem Sie den Überstand nach der Zentrifugation in ein neues Zentrifugenröhrchen überführen.)
- Übertragen Sie die im vorherigen Schritt erhaltene Flüssigkeit vorsichtig in ein neues Zentrifugenröhrchen.
(Notiz: Etwa 500 Es können μl Flüssigkeit übertragen werden; für einige Arten, es kann weniger sein als 500 μl.)
- Fügen Sie dem Lysat ein gleiches Volumen Chloroform hinzu und drehen Sie es vorsichtig um, um es zu mischen.
(Notiz: Zum Beispiel, hinzufügen 500 μl Chloroform, falls vorhanden 500 μl Lysat. Wenn das Lysatvolumen kleiner ist als 500 μl, Passen Sie die Chloroformmenge entsprechend an.)
- Zentrifuge bei 12,000 U/min für 10 Protokoll.
- Übertragen Sie den Überstand vorsichtig in ein neues Zentrifugenröhrchen (etwa 500 μl).
- Fügen Sie eine gleiche Menge hinzu Reagenzpuffer C und ein gleiches Volumen wasserfreies Ethanol zum Lysat, und mischen.
(Zum Beispiel, wenn Sie hinzufügen 450 μl Reagenzpuffer C, dann hinzufügen 450 μl wasserfreies Ethanol. Wenn das Lysatvolumen kleiner ist als 450 μl, Reduzieren Sie die Menge an Reagenzpuffer C proportional. Bei der Zugabe von Reagenzpuffer C kommt es zu einer gewissen Ausfällung, Dies hat jedoch keinen Einfluss auf nachfolgende Experimente.)
- Übertragen Sie die erhaltene Flüssigkeit auf eine DNA-Reinigungssäule (Bausatz), etwa 650-700 jedes Mal μl. Zentrifuge bei über 8,000 U/min für 1 Minute, Entsorgen Sie den gesammelten Abfall, und setzen Sie das Sammelröhrchen für den nächsten Schritt wieder in die Reinigungssäule ein.
- Schritt wiederholen 11, Zugabe der restlichen Flüssigkeit zur DNA-Reinigungssäule (Bausatz) und bei über zentrifugieren 8,000 U/min für 1 Minute. Entsorgen Sie den Abfall und das Sammelröhrchen.
- Platzieren Sie die DNA-Reinigungssäule (Bausatz) in ein neues Sammelröhrchen geben, hinzufügen 300 μl von Waschen Puffer 1, Zentrifuge bei über 8,000 U/min für 1 Minute, Entsorgen Sie den Abfall, und setzen Sie die DNA-Reinigungssäule wieder ein (Bausatz) in die Röhre für den nächsten Schritt.
(Notiz: Stellen Sie sicher, dass wasserfreies Ethanol hinzugefügt wurde Waschen Puffer 1.)
- Hinzufügen 500 μl Spülpuffer 1 zur DNA-Reinigungssäule (Bausatz), Zentrifuge bei 14,000 U/min (20,000×g) für 2 Protokoll, Bei einer trockeneren Membran kann die Zentrifugationszeit etwas verlängert werden.
- Platzieren Sie die DNA-Reinigungssäule (Bausatz) in ein neues Zentrifugenröhrchen füllen, offen, und bei 65°C erhitzen 2 Protokoll. Dieser Schritt kann verlängert werden, um Ethanol so weit wie möglich zu verdampfen und zu verhindern, dass restliches Ethanol die nachgeschalteten Experimente beeinträchtigt.
- Tropfen 100 μl Elutionspufferauf die Membran, Zentrifuge bei 12,000 U/min für 2 Protokoll.
(Notiz: 1. Eluierende DNA mit 50 μl Elutionspuffer kann die DNA-Konzentration erhöhen, aber die Gesamt-DNA-Ausbeute verringern. 2. Das Eluat kann für eine zweite Elution erneut auf die DNA-Reinigungssäule aufgetragen werden, Zentrifuge bei 12,000 U/min für 2 Minuten zum Sammeln, was die DNA-Ausbeute verbessern kann.)












Bewertungen
Es gibt noch keine Bewertungen.