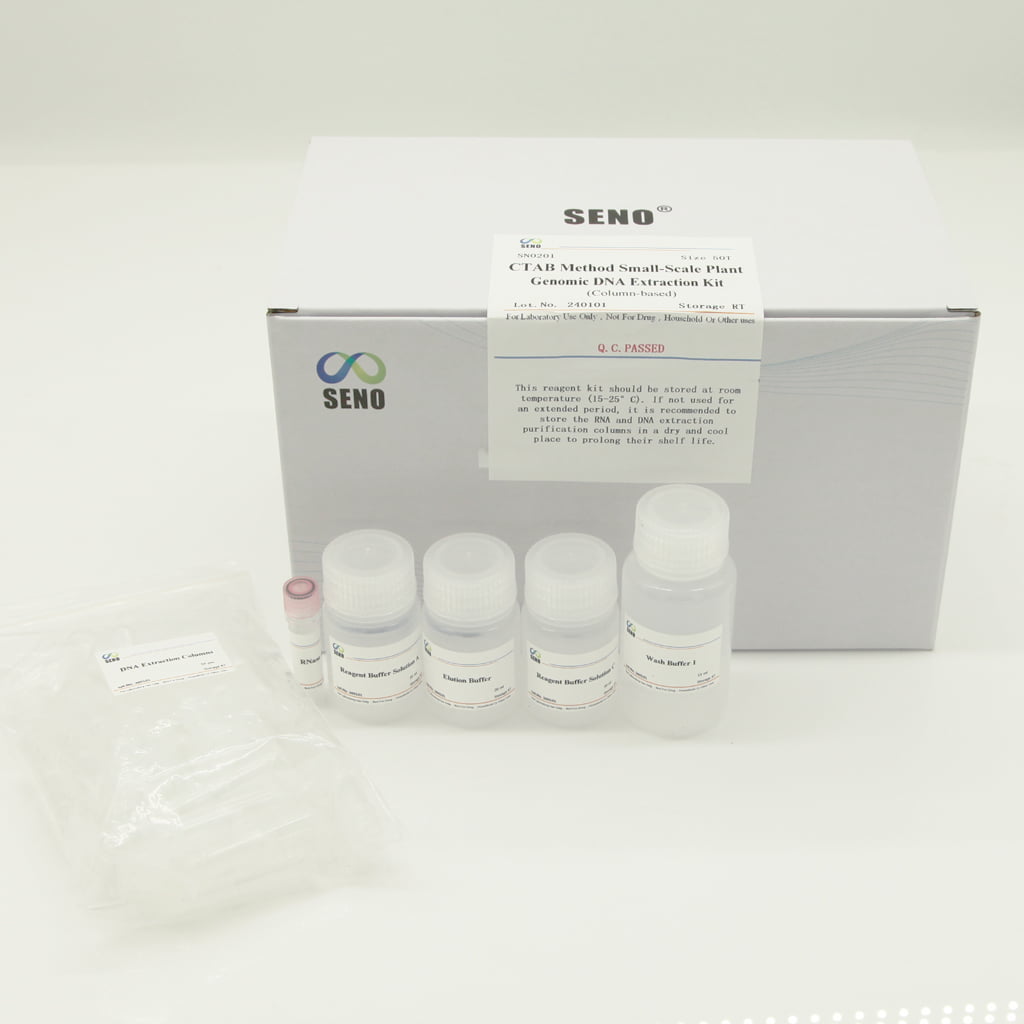
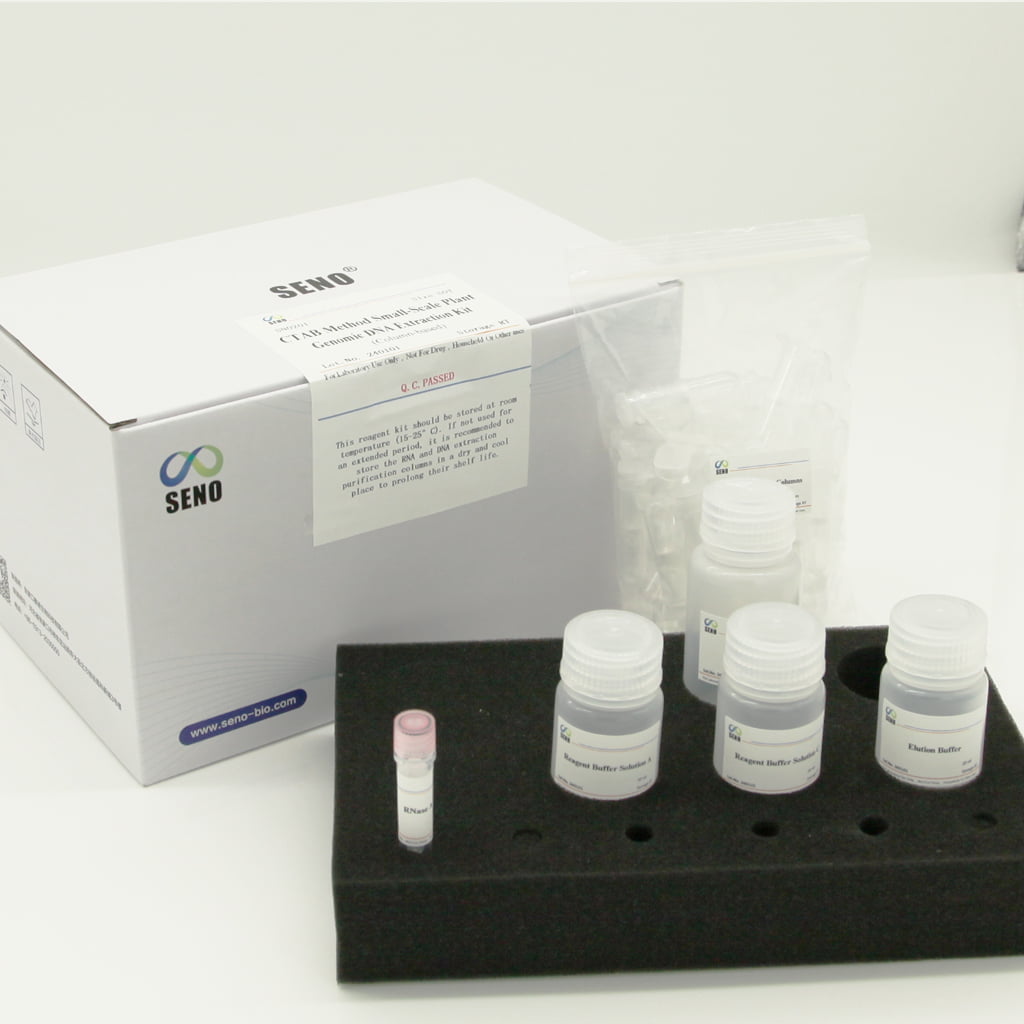
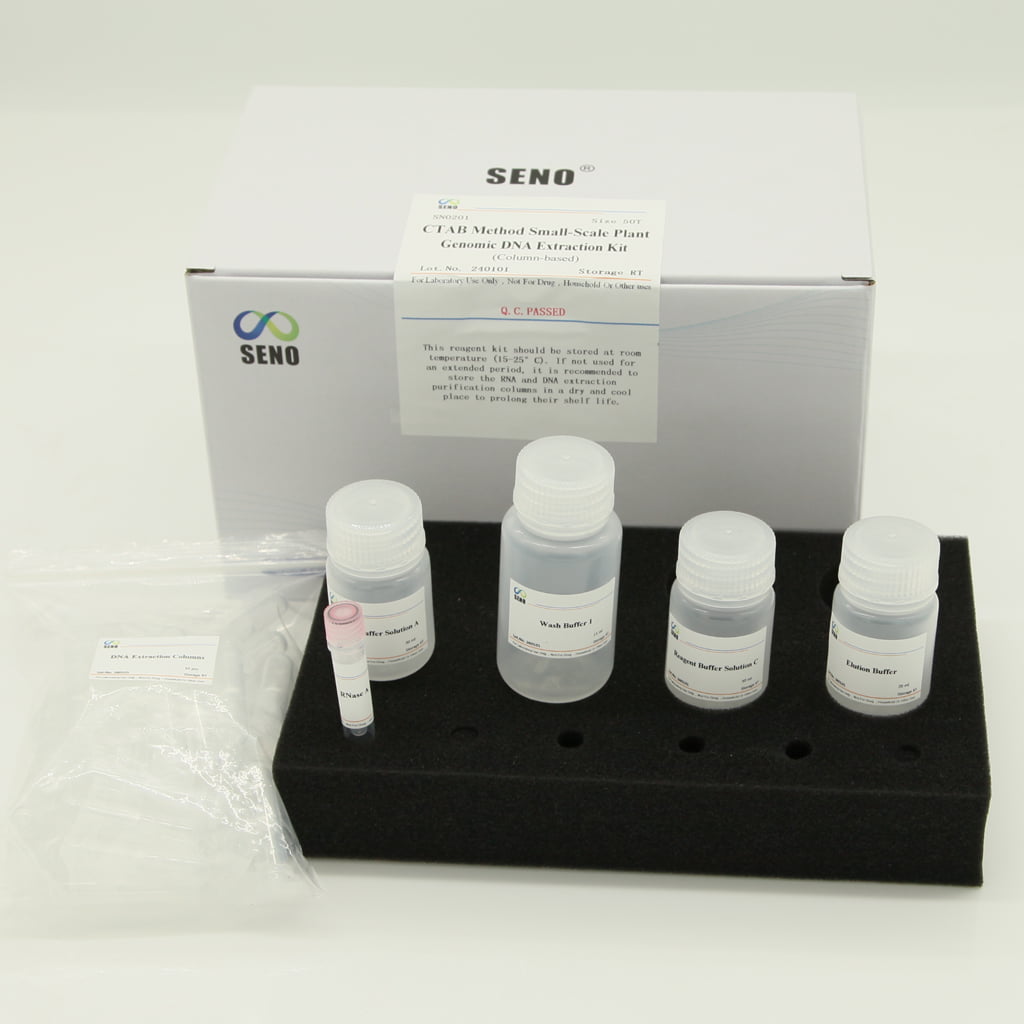
- ส่วนประกอบของชุดรีเอเจนต์
| ข้อมูลจำเพาะ | 50ต | 100ต |
| แมว. เลขที่. | SN0201 | SN0202 |
| คอลัมน์สกัดดีเอ็นเอ (ชุด) | 50 (ชุด) | 100 (ชุด) |
| สารละลายบัฟเฟอร์รีเอเจนต์ A | 30 มล | 2 × 30 มล |
| สารละลายบัฟเฟอร์รีเอเจนต์ C | 30 มล | 2 × 30 มล |
| ล้างบัฟเฟอร์ 1 | 15 มล | 2 × 15 มล |
| อาร์เนส เอ | 1มล | 1มล |
| บัฟเฟอร์การชะล้าง | 20 มล | 20 มล |
| คู่มือการใช้งาน | 1 | 1 |
- พื้นที่จัดเก็บ
ชุดนี้ควรเก็บไว้ที่อุณหภูมิห้อง (15-25℃) ในที่แห้งและสามารถเก็บไว้ได้ 12 เดือน. คอลัมน์การทำให้บริสุทธิ์จากการสกัด DNA สามารถเก็บไว้ในสภาพแวดล้อมที่เย็นและแห้งได้นานถึง 1 ปี. RNase A มีสารกันบูดและสามารถขนส่งได้ที่อุณหภูมิห้อง, แต่สำหรับการจัดเก็บระยะยาว, ควรเก็บไว้ที่ -20 ℃.
- คำแนะนำในการใช้ชุดรีเอเจนต์
3.1 ชุดนี้มีวัตถุประสงค์เพื่อการวิจัยอณูชีววิทยา และไม่ควรใช้สำหรับการวินิจฉัยหรือการรักษาโรค.
3.2 ส่วนประกอบบางอย่างในชุดมีสารระคายเคือง; ขอแนะนำให้ใช้ความระมัดระวังที่จำเป็น (เช่น การสวมชุดป้องกันและแว่นตา).
3.3 การใช้ชุดอุปกรณ์นี้ต้องใช้อุปกรณ์เพิ่มเติม เช่น เครื่องหมุนเหวี่ยงความเร็วสูง, อ่างอาบน้ำ (อาบน้ำโลหะ), เครื่องผสมน้ำวน, เอทานอลปราศจากน้ำ, ไนโตรเจนเหลว, คลอโรฟอร์ม, น้ำปราศจากไอออนฆ่าเชื้อ, และท่ออีพี.
- ข้อมูลเบื้องต้นเกี่ยวกับชุดรีเอเจนต์
ที่ CTAB-โรงงานพื้นฐาน การทำให้บริสุทธิ์ของดีเอ็นเอ Kit นำเสนอวิธี CTAB ที่ได้รับการปรับปรุงสำหรับการทำให้ DNA บริสุทธิ์, ใช้บัฟเฟอร์การจับเฉพาะที่ตกตะกอน DNA ได้อย่างมีประสิทธิภาพ, และต่อมารวบรวม DNA ที่มีความบริสุทธิ์สูงผ่านคอลัมน์ดูดซับ.
ชุดนี้ใช้กันอย่างแพร่หลายสำหรับเนื้อเยื่อพืชและเชื้อรา, สามารถสกัด DNA ทั้งหมดจากตัวอย่างภายในได้ 2 ชั่วโมง (รวมถึงไมโตคอนเดรีย DNA และคลอโรพลาส DNA). DNA ที่สกัดออกมาสามารถนำมาใช้โดยตรงสำหรับการทดลองขั้นปลายน้ำ เช่น พีซีอาร์, การซับใต้, และคนอื่น ๆ.
- หลักและวิธีการทดลอง
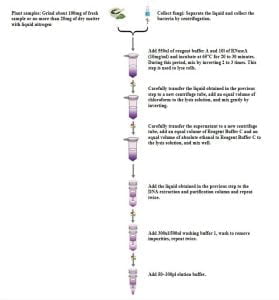
- กระบวนการสกัด
ข้อควรระวังก่อนเริ่มการทดลอง:
- รีเอเจนต์บัฟเฟอร์ A และ C อาจตกตะกอนภายใต้สภาวะอุณหภูมิต่ำ. แนะนำให้อุ่นที่อุณหภูมิ 65°C เป็นเวลา 5 นาทีและใช้หลังจากตะกอนละลายหมดแล้ว.
- ล้างกันชน 1 ควรเติมเอทานอลแบบแอนไฮดรัสตามจำนวนที่กำหนดตามที่ระบุไว้บนฉลากขวด. ทำเครื่องหมายฉลากเมื่อเติมเอธานอลแล้ว.
- บัฟเฟอร์การชะล้างคือ a 0.1x โซลูชัน TEที่มี EDTA น้อยที่สุด. หาก EDTA ส่งผลต่อการทดลองครั้งต่อไป, แนะนำให้ใช้น้ำปราศจากไอออนที่ผ่านการฆ่าเชื้อแทนบัฟเฟอร์การชะล้าง.
- การจัดการตัวอย่าง:
- การรวบรวมและจัดเก็บวัสดุ:
วัสดุที่รวบรวมสดใหม่, หากไม่ได้ใช้ทันที, ควรใส่ในไนโตรเจนเหลวและเก็บไว้ที่อุณหภูมิ -80°C ในที่สุด. วัสดุแห้งสามารถเก็บไว้ที่อุณหภูมิห้องได้.
- ถ้าเป็นไปได้, เก็บวัสดุสดเนื่องจากมีโพลีแซ็กคาไรด์และโพลีฟีนอลน้อยลง.
- เมื่อเก็บเชื้อราจากการเพาะเลี้ยงของเหลว, แยกของเหลวด้วยการหมุนเหวี่ยงและรวบรวมตัวเชื้อรา.
- บดไปรอบๆ 100 ตัวอย่างสด มก. หรือไม่เกิน 20 มิลลิกรัมของวัสดุแห้งโดยใช้ไนโตรเจนเหลว.
(บันทึก: ปริมาณตัวอย่างที่แตกต่างกันอาจต้องมีการปรับให้เหมาะสมผ่านการทดลองเบื้องต้นก่อนใช้งาน.)
- เพิ่ม 550 μl ของบัฟเฟอร์รีเอเจนต์ A และ 10 μl ของ RNase A (10 มก./มล) เพื่อให้แน่ใจว่าไม่มีก้อนเนื้อเยื่อในตัวอย่างภาคพื้นดิน. การจับตัวเป็นก้อนของเนื้อเยื่อนั้นแยกออกได้ยากและอาจส่งผลให้ปริมาณดีเอ็นเอลดลง. อย่าผสม รีเอเจนต์บัฟเฟอร์ A และ RNase Aก่อนใช้งาน.
- ฟักที่อุณหภูมิ 65°C เป็นเวลา 20-30 นาที, ค่อยๆ พลิกกลับ 2-3 ครั้ง. ขั้นตอนนี้มีไว้สำหรับการสลายเซลล์.
- ปั่นแยกไลซีนเพื่อ 5 นาทีที่ 14,000 รอบต่อนาที (20,000×ก).
(บันทึก: วัสดุจากพืชบางชนิดอาจมีสารเหนียวมากในขั้นตอนนี้, ซึ่งสามารถตัด DNA ในขั้นตอนต่อไปได้. ตามหลักการแล้ว, กำจัดสารเหล่านี้โดยการถ่ายโอนส่วนเหนือตะกอนไปยังหลอดหมุนเหวี่ยงใหม่หลังจากการปั่นแยก.)
- ถ่ายของเหลวที่ได้รับในขั้นตอนก่อนหน้าไปยังหลอดหมุนเหวี่ยงใหม่อย่างระมัดระวัง.
(บันทึก: ประมาณ 500 สามารถถ่ายโอนของเหลว µl ได้; สำหรับบางชนิด, มันอาจจะน้อยกว่า 500 ไมโครลิตร.)
- เติมคลอโรฟอร์มในปริมาณที่เท่ากันลงในไลเซตและค่อยๆ กลับด้านเพื่อผสม.
(บันทึก: ตัวอย่างเช่น, เพิ่ม 500 ไมโครลิตรของคลอโรฟอร์มถ้าคุณมี 500 ไมโครลิตรของไลเซต. หากปริมาตรไลซีนน้อยกว่า 500 ไมโครลิตร, ปรับปริมาตรคลอโรฟอร์มให้เหมาะสม.)
- เครื่องปั่นแยกที่ 12,000 รอบต่อนาทีสำหรับ 10 นาที.
- ถ่ายเทส่วนลอยเหนือตะกอนไปยังหลอดหมุนเหวี่ยงใหม่อย่างระมัดระวัง (ประมาณ 500 ไมโครลิตร).
- เพิ่มปริมาณที่เท่ากันของ รีเอเจนต์บัฟเฟอร์ C และเอธานอลแอนไฮดรัสในปริมาณเท่ากันกับไลซีน, และผสม.
(ตัวอย่างเช่น, ถ้าคุณเพิ่ม 450 ไมโครลิตรของบัฟเฟอร์รีเอเจนต์ C, จากนั้นเพิ่ม 450 เอทานอลปราศจากน้ำ ไมโครลิตร. หากปริมาตรไลซีนน้อยกว่า 450 ไมโครลิตร, ลดปริมาณบัฟเฟอร์รีเอเจนต์ C ตามสัดส่วน. การตกตะกอนบางส่วนจะเกิดขึ้นเมื่อมีการเติมบัฟเฟอร์รีเอเจนต์ C, แต่จะไม่ส่งผลต่อการทดลองครั้งต่อไป.)
- ถ่ายโอนของเหลวที่ได้รับไปยังคอลัมน์การทำให้บริสุทธิ์ของ DNA (ชุด), ประมาณ 650-700 ไมโครลิตรในแต่ละครั้ง. ปั่นแยกที่มากกว่า 8,000 รอบต่อนาทีสำหรับ 1 นาที, ทิ้งขยะที่รวบรวมไว้, และใส่ท่อรวบรวมกลับเข้าไปในคอลัมน์การทำให้บริสุทธิ์สำหรับขั้นตอนต่อไป.
- ทำซ้ำขั้นตอน 11, เติมของเหลวที่เหลือลงในคอลัมน์การทำให้บริสุทธิ์ของ DNA (ชุด) และหมุนเหวี่ยงที่ด้านบน 8,000 รอบต่อนาทีสำหรับ 1 นาที. ทิ้งของเสียและท่อรวบรวม.
- วางคอลัมน์การทำให้บริสุทธิ์ DNA (ชุด) ลงในหลอดสะสมใหม่, เพิ่ม 300 ไมโครลิตรของ ล้าง กันชน 1, ปั่นแยกที่มากกว่า 8,000 รอบต่อนาทีสำหรับ 1 นาที, ทิ้งขยะ, และใส่คอลัมน์การทำให้บริสุทธิ์ DNA กลับเข้าไปใหม่ (ชุด) ลงในท่อเพื่อขั้นตอนต่อไป.
(บันทึก: ตรวจสอบให้แน่ใจว่าได้เติมเอธานอลปราศจากน้ำแล้ว ล้าง กันชน 1.)
- เพิ่ม 500 μl ของบัฟเฟอร์ล้าง 1 ไปยังคอลัมน์การทำให้บริสุทธิ์ของ DNA (ชุด), เครื่องหมุนเหวี่ยงที่ 14,000 รอบต่อนาที (20,000×ก) สำหรับ 2 นาที, ยืดเวลาการปั่นแยกเล็กน้อยสำหรับเมมเบรนแห้ง.
- วางคอลัมน์การทำให้บริสุทธิ์ DNA (ชุด) ลงในหลอดหมุนเหวี่ยงใหม่, เปิด, และตั้งอุณหภูมิไว้ที่ 65°C เป็นเวลา 2 นาที. ขั้นตอนนี้อาจยืดเยื้อออกไปในการระเหยเอธานอลให้มากที่สุดเพื่อป้องกันไม่ให้เอธานอลตกค้างส่งผลต่อการทดลองขั้นปลายน้ำ.
- หยด 100 μl ของบัฟเฟอร์การชะล้างลงบนเมมเบรน, เครื่องหมุนเหวี่ยงที่ 12,000 รอบต่อนาทีสำหรับ 2 นาที.
(บันทึก: 1. การชะล้าง DNA ด้วย 50 บัฟเฟอร์การชะ µl สามารถเพิ่มความเข้มข้นของ DNA แต่ลดผลผลิต DNA ทั้งหมด. 2. สารชะสามารถนำไปใช้ซ้ำกับคอลัมน์การทำให้บริสุทธิ์ของ DNA เพื่อการชะล้างครั้งที่สอง, เครื่องหมุนเหวี่ยงที่ 12,000 รอบต่อนาทีสำหรับ 2 นาทีในการรวบรวม, ซึ่งอาจช่วยเพิ่มผลผลิต DNA.)













รีวิว
ยังไม่มีบทวิจารณ์